Origem do SARS-CoV-21
Ginhas Alexandre Manuel, José Gomes Ngandu, Adriano Bingobingo, Orlis Bárbara Alfonso Loret De Mola
Faculdade de Ciências Agrárias, Universidade José Eduardo dos Santos, Huambo, Angola
Este endereço de email está protegido contra piratas. Necessita ativar o JavaScript para o visualizar.; Este endereço de email está protegido contra piratas. Necessita ativar o JavaScript para o visualizar.; Este endereço de email está protegido contra piratas. Necessita ativar o JavaScript para o visualizar.; Este endereço de email está protegido contra piratas. Necessita ativar o JavaScript para o visualizar.
Introdução
A actual pandemia de coronavírus COVID-19, foi relatada pela primeira vez na cidade de Wuhan, província de Hubei, República Popular da China, em Dezembro de 2020. Apesar dos esforços a nível global para o seu controlo, a mesma propagou-se praticamente por todos os paises, causando até 13 Julho de 2020, cerca de 13 milhões de casos confirmados e mais de 560 000 obitos (OMS, 2020).
A COVID-19 é uma doença causada pelo novo betacoronavírus SARS-CoV-2, que causa sintomas respiratórios agudos, febre, tosse seca e fatiga. Nos casos mais graves de indivíduos imunocomprometidos, a doença pode provocar uma infecção bacteriana secundária, como a pneumonia, insuficiência renal e até, a morte (Pal et al., 2020). O SARS-CoV-2 é similar aos betacoronavirus que causam a síndrome respiratória aguda grave (SARS) e da síndrome respiratória do Médio Oriente (MERS), que causaram mais de 10.000 casos nas últimas duas décadas, com uma taxa de mortalidade de 10% para SARS-CoV e 37% para MERS-CoV (Gillespie et al., 2020; Sohrabia et al., 2020; Ena et al., 2020).
Investigações recentes, apontam que o SARS-CoV-2 tem um reservatório animal, geralmente animais selvagens, podendo transmitir-se aos humanos, pelo que se teoriza que este vírus é zoonótico, com probabilidade de propagar-se entre as espécies (de Wilde et al., 2018). Apesar dos esforços conjuntos de investigação, pouco se sabe sobre o reservatório natural deste vírus.
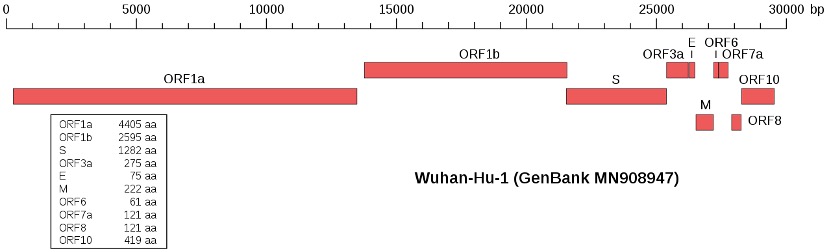
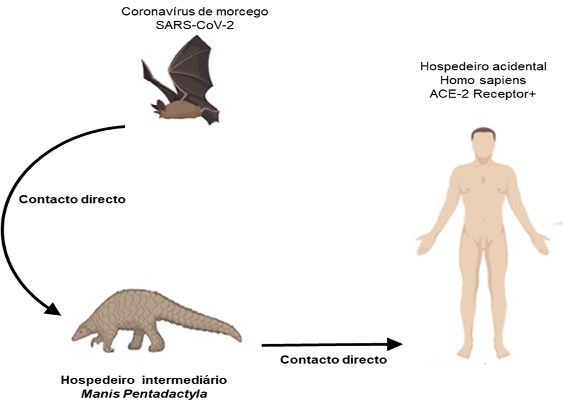
Alguns cientistas acreditam que a doença se originou nos morcegos chineses antes de saltar para um animal intermediário como o pangolim, e a partir daí, encontrar seu caminho nos seres humanos, como se representa na figura 1 (Callaway et al., 2020; Liu et al., 2020; Singh, 2020). Neste sentido Xu et al. (2020) destacam que pesquisas realizadas pela Universidade Agrícola do Sul da China apontam o pangolim como hospedeiro intermediário da SARS-CoV-2. Depois de analisar mais de 1.000 amostras metagenômicas, descobriram que aproximadamente o 70% dos pangolim são positivos para o coronavírus. Além disso, o vírus isolado do pangolim compartilhou 99% de similaridade de sequência com o actual vírus de estirpe humana SARS-CoV-2.
Em contraste, Ji et al. (2020), em um estudo recente sobre possíveis reservatórios do SARS- CoV-2, sugerem que o SARS-CoV-2 é um vírus recombinante derivado de um coronavírus de morcego e de outro coronavírus desconhecido. A recombinação entre as duas espécies de vírus, ocorreu no nível da glicoproteína que reconhece os receptores da superfície celular. Os resultados indicam que algumas cobras da China podem ser o reservatório selvagem mais provável para SARS-CoV-2, dada à similaridade no viés relativo no uso de códons sinônimos em comparação com os de morcego.
Actualmente, a preocupação global se baseia em estudos de análises filogenéticos que possam esclarecer se os animais domésticos e peridomésticos podem servir de hospedeiros intermediários ou facilitar a recombinação do vírus, servindo de hospedeiros de amplificação.
Esta revisão da literatura aborda aspectos relevantes que permitiram responder as seguintes questões de investigação:
- O SARS-CoV-2 pode transmitir-se de humanos a outras espécies animais?
- Os animais domésticos, depois de infectados, podem tornar-se reservatórios do virus SARS-CoV-2?
Risco da transmissão do SARS-CoV-2 de humanos para animais domésticos e de estimação
A Organização Mundial da Saúde Animal (OIE) e a Organização Mundial da Saúde (OMS), para garantir a saúde pública e animal, recomendam que as pessoas infectadas com a COVID-19, devem reforçar as medidas de higiene, e evitar o contacto com seus animais de estimação, enquanto estiverem doentes, salientando, que existem evidências de que os pacientes com COVID-19 podem infestar os seus animais de companhia.
Pois, o primeiro caso de transmissão de humano para animal, foi relatado por um porta-voz do Departamento de Agricultura, Pesca e Conservação (AFCD) de Hong Kong. Tratou-se de um cão de estimação, que testou positivo ao vírus SARS-CoV-2, enquanto convivia com seu proprietario infectado com o vírus da COVID-19. Acredita-se que a infecção do cão, tenha sido possivelmente, através do seu proprietário (AFCD, 2020). Esta mesma fonte, informa que testaram 17 cães e oito gatos, de famílias com casos confirmados de COVID-19, ou de pessoas próximas com pacientes confirmados, e apenas dois cães testaram positivo para o vírus SARS-CoV-2.
Segundo a informação do Centro de controlo e prevenção de doenças (CDC), na cidade de Nova York, EUA, um tigre com uma doença respiratória, em um zoológico, testou positivo ao vírus que causa o COVID-19. As amostras do tigre foram coletadas, depois que vários leões e tigres, no referido zoológico, mostraram sinais de doença respiratória. As autoridades de saúde pública acreditam que os felinos tenham contraido a doença, por estarem expostos a um funcionário do zoológico, infectado com o vírus SARS-CoV-2 (CDC, 2020).
Por outro lado, Chen et al. (2015) investigaram diferentes animais domésticos (porcos, galinhas, cães, patos, gatos e gado) que convivem em próximo contacto com o ser humano, correndo o risco de serem infectados por SARS-CoV e observaram que o SARS-CoV humano, pode infectar mamíferos domésticos, em particular o porco, e que provavelmente a fonte da transmissão de SARS-CoV, poderia ser alimentação contaminada, visto que os mesmos eram alimentados com restos provenientes de restaurantes localizados nas cidades. Concluíram que, a vigilância permanente e as investigações em animais, especialmente durante e após um surto de SARS, poderiam levar a uma melhor compreensão e capacidade de controlar os reservatórios de animais naturais desta doença e impedir eventos de transmissão inter-espécies.
Temmam et al. (2020), investigadores do Instituto Pasteur, de Paris, partindo do conhecimento sobre a capacidade do SARS-CoV-2 de infectar alguns animais domésticos, testaram 21 animais (nove gatos e 12 cães) que conviviam com os seus proprietarios que faziam parte de uma comunidade veterinária de 20 alunos, com a finalidade de estudar a ausência de infecção por SARS-CoV-2 em gatos e cães, que mantinham contato próximo com um grupo de pacientes com COVID-19. Os autores concluíram que, embora alguns animais de estimação apresentassem vários sinais clínicos, indicativos de infecção por coronavírus, nenhum animal testou positivo para o SARS-CoV-2 por RT-PCR e nenhum anticorpo foi detectado no sangue, usando um ensaio de imunoprecipitação. Os mesmos, ressaltam que os resultados encontrados, podem servir para melhor avaliação da gama hospedeira de SARS-CoV-2 em ambiente natural e em condições de exposição.
Risco da transmissão do SARS-CoV-2 de animais domésticos para humanos.
A Associação Mundial de Clínicos Veterinários de Pequenos Animais (WSAVA), num documento informativo, destaca, que até o momento, não há evidências, que apontam um hospedeiro animal específico, como reservatório do vírus SARS-CoV-2. Porém, estão em curso várias investigações, para maior esclarecimento do assunto (WSAVA, 2020).
No mesmo sentido, a Organização Mundial da Saúde Animal (OIE) e a Organização Mundial da Saúde (OMS), salientam, que não há dados disponíveis, que confirmam que um cão, um gato ou qualquer animal de estimação, possa transmitir o COVID-19. Logo, por se tratar de uma temática em rápida evolução, as organizações em causa, comprometem-se a disponibilizarem informações atualizadas, no seu site, (spread of newly emerged coronaviruses) à medida que os diferentes grupos de investigação, forem produzindo novos conhecimentos sobre o assunto (OIE, 2020).
Shi et al. (2020), no estudo sobre susceptibilidade de furões, gatos, cães e diferentes animais domésticos ao SARS-CoV-2, infestaram intranasalmente os animais e alojaram, na mesma jaula, animais infectados com os seus parceiros não infectados e, para monitorar a transmissão do vírus, foram colectadas amostras intranasais e rectais. Os autores descobriram que, o SARS-CoV-2, se replica eficientemente em furões e gatos, se replica com dificuldade em cães e gado e não se replica em porcos, galinhas e patos. Os resultados encontrados, indicam que o SARS-CoV-2, pode-se replicar no trato respiratório superior de furões, por até oito dias, sem causar doença grave ou morte. O estudo demonstrou também, que, um em cada três gatos expostos ao vírus (gatos infectados) foi infectado, revelando que, a transmissão intra-espécies, (em gato) foi por via de gotículas respiratória. O estudo fornece informações importantes sobre os reservatórios animais de SARS-CoV-2.
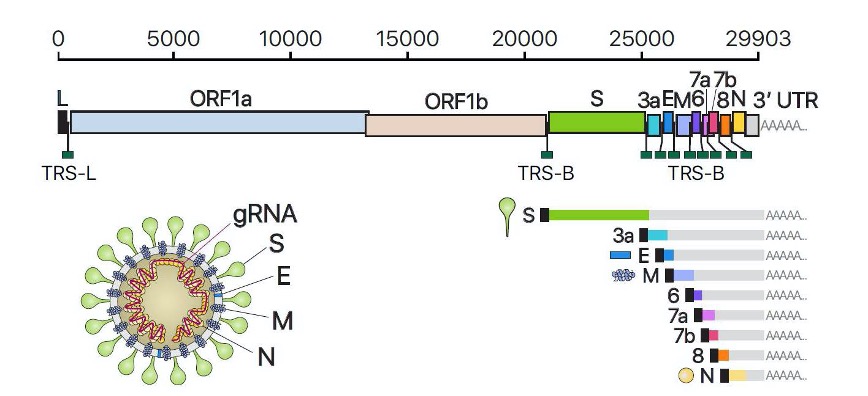
Rodrigues et al. (2020), no trabalho Particularidades da infecção por diferentes estirpes de coronavírus em animais domésticos e de produção, afirmaram que alguns casos isolados de COVID-19, foram confirmados em alguns animais (cães e felinos), ressaltando que várias pessoas que possuem animais domésticos temem pela contaminação. Os autores destacam que a transmissão ocorre de humano a humano, e que os humanos podem contaminar os animais, porém não há evidência que animais contaminem os humanos, como se mostra na Figura 2.
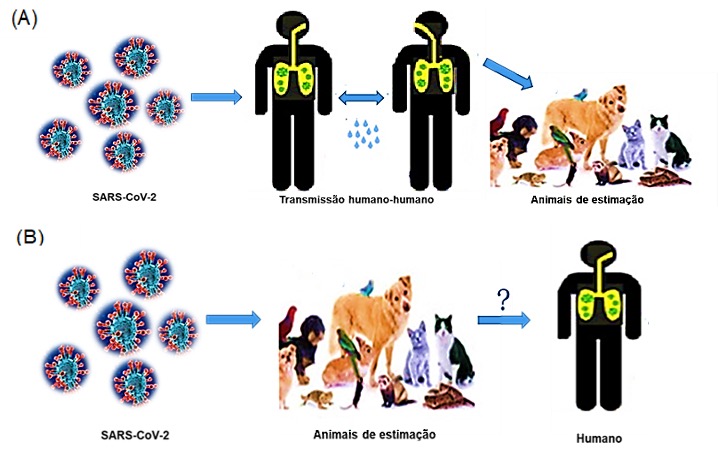
Figura 2. Transmissão do vírus SARS-CoV-2. Se representa com a letra (A) a transmissão de humanos para animais; e com a letra (B) a transmissão de animais para humanos. (Figura adaptada de Perveen, et al., 2020).
Zhang et al. (2020) investigaram a infecção por SARS-CoV-2 em 102 gatos de estimação, na cidade de Wuhan, durante o surto de COVID-19. Os resultados revelaram uma prevalência 14,7% de gatos infestados. Os autores deduzem que, este fenómeno também pode estar a ocorrer em outras regiões com ocorrência da COVID-19, pelo que, os mesmos, alertam para que se executem acções no sentido de manter-se distância entre humanos e animais de companhia, como gatos e cães, e que se estreitem medidas de higiene e se programem também quarentenas para os animais infectados. O estudo apontou o risco de que os gatos possam estar envolvidos na transmissão do SARS-CoV-2.
Goumenou et al. (2020), no artigo publicado no jornal Molecular Medicine Reports, em relação a possível transmissão da COVID-19, através de cães, na região Norte de Itália, manifestam, baseando-se na literatura consultada, que não existe até o momento, evidências de que os cães possam propagar a COVID‑19, para outros animais, incluindo humanos. Ainda assim, os autores, argumentam com dados científicos e factos ocorridos na Itália, durante a avalanche numérica causada pela pandemia, à necessidade de reconsiderar a possibilidade de que os cães sejam hospedeiros intermediários, e tenham contribuído na propagação da COVID-19, na região norte do pais.
Conclusões
Os dados existentes até o momento demostram que a contaminação de humanos para animais é possível. Pelo que, os pacientes com COVID-19, devem reforçar as medidas de higiene, e evitar o contacto com os seus animais de companhia. Até o preciso momento, não existem dados científicos disponíveis, que confirmem ou excluem a possibilidade da propagação do vírus SARS-CoV-2, de animal para humano. porém, estão em curso várias investigações, para maior esclarecimento do assunto. Por outro lado, grande parte dos trabalhos realizados, sobretudo os experimentais, acarreta uma margem de erro considerável, por vários factores, entre os quais, o curto período de tempo de realização dos mesmos, o reduzido tamanho de amostras e as condições de estudos.
Referências
AFCD. 2020. New Updates on Low-level of Infection with COVID‑19 in Pet Dog. 2020. Agriculture, Fisheries and Conservation Department (AFCD). https://www.pets.gov.hk/english/highlights/files/New_ Updates_on_Low-level_of_Infection_with_COVID‑19_in_Pet_Dog_0503__eng.p.
Callaway, E. and Cyranoski, D. 2020. Why snakes probably aren’t spreading the new China virus. Nature. 2020. https://www.nature.com/articles/d41586-020-00180-8.
CDC. 2020. Coronavirus Disease 2019 (COVID-19). [Online] 2020. Center for Disease Control and Prevention (CDC).. https://www.cdc.gov/coronavirus/2019-ncov/daily-life-coping/animals.html.
Chen, W. J., Yan, M. H. and Yang, L. et al. 2015. SARS-associated coronavirus transmitted from human to pig. Emerging Infectious Diseases. 2015, Vol. 11, 3, pp. 446-448. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3298239/.
de Wilde, A. H., et al. 2018. Host Factors in Coronavirus Replication. Curr Top Microbiol Immunol. 2018, Vol. 419, pp. 1-42. [PMC free article] [PubMed] [Google Scholar].
Ena, J. and Wenzel, R. P. 2020. Un nuevo coronavirus emerge. Rev Clin Esp. Março 2020, Vol. 220, 2, pp. 115–116. PMID: 32063263.
Gillespie, T. R. and Leendertz, F. H. 2020. COVID-19: protect great apes during human pandemics,. Nature Research Journal. 26 March 2020, Vol. 579. https://www.nature.com/articles/d41586-020-00859-y.
Goumenou, M., Spandidos, D.A. and Tsatsakis, A. 2020. Possibility of transmission through dogs being a contributing factor to the extreme Covid‑19 outbreak in North Italy (2020). Molecular Medicine REPORTS. 2020, Vol. 21, pp. 2293-2295. Doi: 10.3892/mmr.2020.11037.
Ji, W., et al. 2020. Cross‐species transmission of the newly identified coronavirus 2019‐nCoV. Journal of Medical Virology. 2020, Vol. 92, 4. Special Issue: 2019 Novel. https://doi.org/10.1002/jmv.25682.
Liu, P., et al. 2020. Are pangolins the intermediate host of the 2019 novel coronavirus (2019-nCoV)? bioRxiv preprint. 2020. Doi: https://doi.org/10.1101/2020.02.18.954628.
OIE. 2020. OIE: Questions and answers on the 2019 Coronavirus Disease (COVID-19). 2020. World Organisation for Animal Health (OIE). Acessado em 30/04/2020.
OMS. 2020. Coronavirus disease (COVID-19) Pandemic. 2020. Organização Mundial da Saúde (OMS). https://www.who.int/emergencies/diseases/novel-coronavirus-2019.
Pal, M., et al. 2020. Severe Acute Respiratory Syndrome Coronavirus-2 (SARS-CoV-2): An Update. Cureus. 2020, Vol. 12 , 3 e7423. Doi: 10.7759/cureus.7423.
Perveen, S., et al. 2020. Coronavirus nCOVID-19: A pandemic disease and the Saudi precautions: Elsevier, Saudi Pharmaceutical Journal Science, Vol. 28 (7) pp. 888-897.
Rodrigues, K. M. M. T., et al. 2020. Particularidades da infecção por diferentes estirpes de coronavirus em animais domésticos e de produção. Revista Desafios. 2020, Vol. 7. Supl. COVID-19, 2020. Doi: http://dx.doi.org/ 10.20873/uftsuple2020-8952.
Shi, J., et al. 2020. Susceptibility of ferrets, cats, dogs, and different domestic animals to SARS-coronavirus-2. bioRxiv preprint. 2020. Doi: https://doi.org/10.1101/2020.03.30.015347.
Singh, R.B. 2020. Transmission Cycle of SARS CoV 2. StatPearls Publishing LLC. [Online] NCBI, https://www.ncbi.nlm.nih.gov/books/NBK554776/figure/article-52171.image.f4//. Acessado 09/07/ 2020.
Sohrabia, C., et al. 2020. World Health Organization declares global emergency: A review of the 2019 novel coronavirus (COVID-19). International Journal of Surgery. 2020.
Temmam, S., et al. 2020. Absence of SARS-CoV-2 infection in cats and dogs in close contact with a cluster of COVID-19 patients in a veterinary campus. bioRxiv preprint. 2020. Doi: https://doi.org/10.1101/2020.04.07.029090.
WSAVA. 2020. O Novo Coronavírus e Animais de Companhia – Informação para os Membros da WSAVA. 2020. Comitês Científico e de Saúde Única da WSAVA. Documento Informativo https://wsava.org/wp-content/uploads/2020/03/COVID-19_WSAVA-Advisory-Document-Mar-7-2020-Portuguese.
Xu, J., et al. 2020. Systematic Comparison of Two Animal-to-Human Transmitted Human Coronaviruses: SARS-CoV-2 and SARS-CoV. Viruse. MPDI. 2020.
Zhang, Q., et al. 2020. SARS-CoV-2 neutralizing serum antibodies in cats: a serological investigation. bioRxiv. 2020. Doi: https://doi.org/10.1101/2020.04.01.021196.
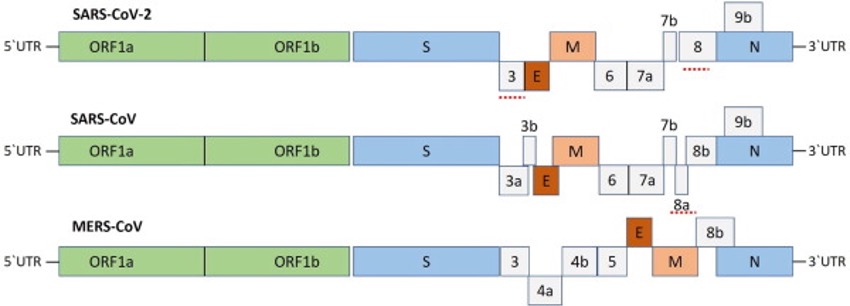
1- Origem do SARS-CoV-2. Representação da possível origem do vírus SARS-CoV-2 a partir de morcegos chineses, tendo como hospedeiro intermediário o pangolim, evoluindo posteriormente, por contacto directo ao homo sapiens. (Figura adaptada de Singh, 2020).